-
Notifications
You must be signed in to change notification settings - Fork 1
PyMOL
filips edited this page Jun 30, 2021
·
60 revisions
- uruchamiamy terminal (Start -> terminal) lub klawiszami ctrl+alt+t
- wpisujemy
pymol
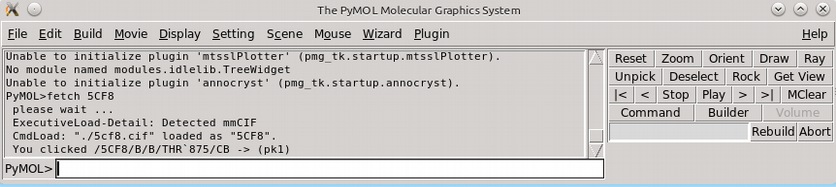
- File - otwieranie, zapisywanie plików i sesji.
- Wizard - czarodzieje, np pomiar odległości i kątów
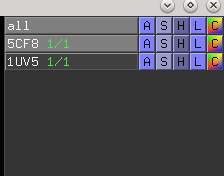
- A - Actions
- S - Show
- H - Hide
- L - Labels - etykiety
- C - Colors
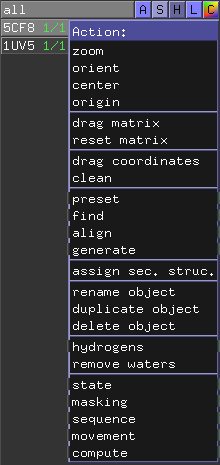
- zoom, orient, center - przybliża, centruje widok na wybranym obiekcie
- preset - predefiniowane widoki, np pretty, publication, ligand sites
- align - alignowanie = wyrównywanie dwóch obiektów, np dwóch białek.
- rename object - zmiana nazwy obiektu
- duplicate object - zduplikowanie obiektu
- delete object - usunięcie obiektu
- hydrogens - manipulacja wodorami - dodanie, usunięcie itp
- remove waters - usunięcie wód (H2O)
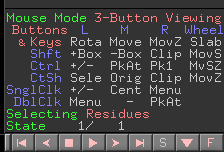
- Selecting - co będzie zaznaczane podczas kliknięcia: reszty (residues), atoms (atomy), chains (łańcuchy)...
- S - pokaż sekwencję (interaktywne!)
- F Full screen
| co napisać | co to robi |
|---|---|
fetch 5CF8 |
Pobiera z bazy PDB strukturę o podanym kodzie |
set_name old_name, new_name |
Zmiana nazwy obiektu |
| co napisać | co to robi |
|---|---|
bg_color white |
kolor tła - biały |
set transparency, 0.5 |
przezroczystość |
ray |
raytracing |
set grid_mode,1 set grid_mode,0
|
włącza/wyłącza podział ekranu na części |
| co napisać | co to robi |
|---|---|
png |
Zapisuje to co widać jako plik graficzny png |
h_add |
Dodaje wodory do struktur |
distance wodorowe, receptor, ligand, mode=2 |
Pokazuje wiązania wodorowe między receptorem a ligandem |
show_bumps |
Pokazuje nałożenia steryczne reszt (clashes) wiki |
| co napisać | co to robi |
|---|---|
cartoon loop |
Pocienia cartoon (więcej przykładów) |
label n. CA, "%s%s" % (resn, resi) |
dodanie etykiet do aminokwasów |
set stereo_mode, 10stereo
|
obraz 3D do oglądania w okularach niebiesko-różnowych (inne przykłady) Aby wyjść ze stereo mode, wpisz: stereo off
|
select nowa nazwa, SELEKTOR
| SELEKTOR | co znaczy |
|---|---|
symbol |
atomy o symbolu, np select polar, symbol n+o
|
resn |
nazwy reszt aminokwasowych, np: select aminokwasy, resn GLY+ALAlub innych nazw, np woda: select woda, resn HOH
|
resi |
Identyfikator (numer) aminokwasu, np: select reszty, resi 210+222+311albo zakres: select reszty, resi 210-311
|
chain |
Łańcuch, np select lancuchB, chain B
|
Inne
import findseq
findseq FATEW, protein # szuka sekwencji FATEW w protein
- Zaznaczenie aminokwasów hydrofobowych:
select hydrophobes, resn ALA+GLY+VAL+ILE+LEU+PHE+MET+TYR+TRP - Zaznaczenie atomów w okolicy ligandu oprócz innych ligandów (heteroatomów):
select okolice, (ligand around 8) and not het - lepsze - bo zaznacza całe reszty:
select br. all within 8 of ligand - tylko w białku:
select okolica, br. bialko within 5 of ligand
extract lig, organic
extract prot, polymer
show surface, prot within 8 of lig
set two_sided_lighting
set transparency, 0.5
show sticks, lig
orient lig
set surface_color, white
select near_ten, lig around 5
select complete_res, br. near_ten
# show as sticks
# set cartoon_fancy_helices, 0
# set cartoon_transparency, 0.5
# set ray_trace_mode, 3
# wywala wszystko co jest dalej niż 10 A od objektu
remove * beyond 10 of objekt
# rename object
set_name old_name, new_name
# cienkie linie
set line_width, 0.5
# label size and stuff, see: https://pymol.org/dokuwiki/?id=setting:label
# 1. - label with labelling wizard
set label_size, 36
set label_bg_outline, true
set label_bg_color, silver
set label_connector, true
# All in one 1:
set label_size, 36; set label_bg_outline, true; set label_bg_color, silver; set label_connector, true
## All in one 2:
set label_bg_outline, true; set label_bg_color, silver; set label_connector, true; set label_connector_color, silver; set label_connector_mode, 2
label (sele), "%s-%s" % (resn, resi)
# przeźroczyste tło w renderach
set ray_opaque_background, off
# rozmiar sfery (np woda)
set sphere_scale, 0.1
# rozczłonkowanie wszytskich states - w tę i we wtę
set all_states, 1
set all_states, 0
# sates
create pose_213, all+ref, 213
set state, 213, pose_213
# split states first and last:
split_states sele, 100, 101
# np dla cartoons, np (liczby 0 - default, 1,2,3) - rysunkowe, czarno-białe itp.
set ray_trace_mode, 3Loop over all objects and do something:
[print(x) for x in cmd.get_object_list('Filip*') ]
[cmd.align(x, 'ligand') for x in cmd.get_object_list('mm*') ]
# small molecules, with psico
# https://pymolwiki.org/index.php/Mcsalign
# https://pymolwiki.org/index.php/Psico
[cmd.mcsalign(x, 'ligand') for x in cmd.get_object_list('mm*') ]
[cmd.enable(x) for x in cmd.get_object_list('Filip*') ]
[cmd.extract("%s-ligand" % (x), "organic and %s" % (x) ) for x in cmd.get_object_list('*M') ]
[cmd.pseudoatom("label--"+x) for x in cmd.get_object_list('*M') ]
[cmd.label(x, '"%s" ' % ( x.upper()[:4] ) ) for x in cmd.get_object_list('label--*') ]Simple script:
for x in cmd.get_object_list('Filip*'):
print(x)
cmd.enable(x);
cmd.ray()
cmd.png("%s.png" % (x) )
cmd.disable(x);# podpisanie całej struktury
pseudoatom forLabel
label forLabel, "This Protein is a Carbohydrate Active Enzyme"
set label_color, black
mplaymstopmpng- bash:
mencoder "mf://*.png" -mf type=png:fps=15 -ovc lavc -o output.avi
Computer Aided Drug Design @ Politechnika Warszawska, Filip Stefaniak